用Python从头实现Needleman-Wunsch序列比对算法
本文将介绍生物信息学中一个基本的序列比对算法,Needleman-Wunsch比对算法(以下简称NW算法),并展示如何编写一个简单的Python程序来实现该算法。全文分为上下两个部分:上篇介绍并阐释NW算法的原理,下篇展示如何用Python实现。如果你对该算法不陌生并且仅对实现方法感兴趣,那么可以直接跳到下篇。
——————————————————— 上篇 ————————————————————
序列比对是生物信息学领域的经典问题,出现了很多算法,也衍生出了一大批生物信息学工具,有些已经逐渐淡出历史舞台,有些依然被频繁使用,比如MUSCLE,BLAST,BWA,Bowtie2等。
两序列间的比对(“一对一”)是序列比对话题中最基本的问题,也是本文将要探讨的主题,更复杂的比对问题都可以由此衍生而来,比如,“多对多”:多重序列比对;“一对多”:目标序列对数据库的比对;“多对一”:大量二代测序短序列比对到人类基因组上。
两序列比对,从解决问题的方向上来说,可下分为全局比对(Global Alignment)和局部比对(Local Alignment)两种。前者试图将两条序列从头到尾整体对齐,而后者则只是针对两条序列中高度相似的局部区域进行比对。这两种方式都各有特点,局部比对的意义在于,在实际研究中,我们往往更关注序列间高度保守的区域,因为这暗示着重要的生物学功能。同样的两条序列,采用全局比对和局部比对都可以,但得到的结果可能差别很大:

NW算法就是一种经典的全局比对算法,由Saul B. Needleman和Christian D. Wunsch于1970年首次提出,当时是为了解决蛋白序列的比对问题,至今依然在使用。值得一提的是,同时期涌现了许多重要的比对算法,比如Smith-Waterman算法。但NW算法是最早的、也是最经典的一个,所以你可能会在很多生物信息学专著中见到它,比如:

下面开始介绍NW算法。
第一步,定一个打分策略。这是基础, 定义了两个碱基在各种比对情况下的分值(包括gap),我们这里将会用到一个简单的:
若是两个碱基一样,即完美匹配,即 +1 分
若是两个碱基不一样,即错配,则 -1 分
若是在任一条链上开一个gap,则 -1 分我们用 θ(a, b)函数来表示碱基 a 和 b 之间的打分。
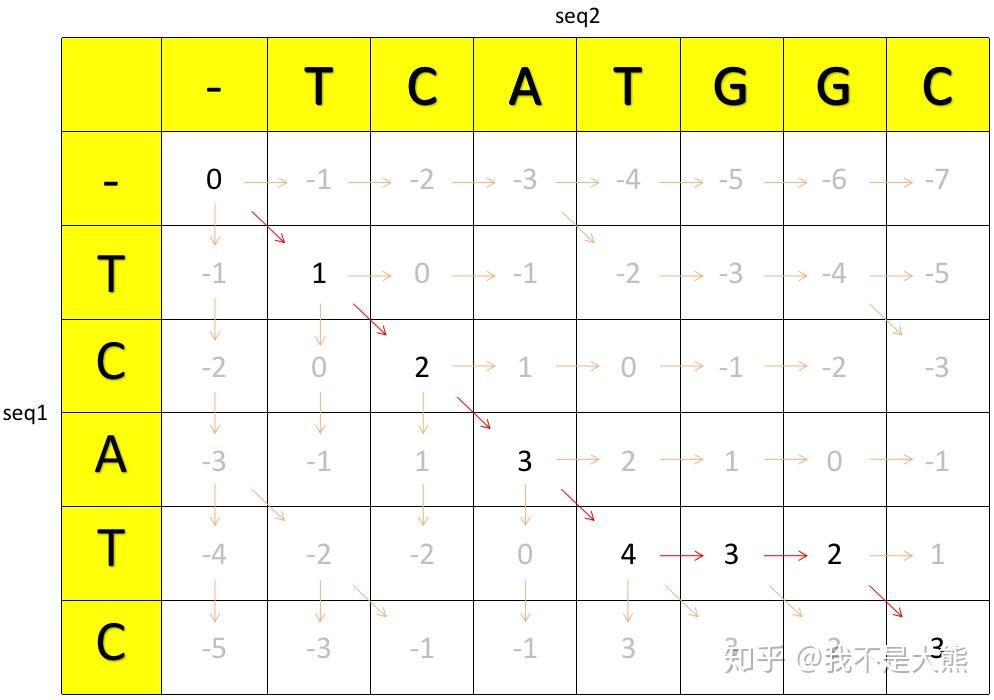
第二步,计算分值矩阵。分值矩阵的概念是NW算法的重点,矩阵由格子组成,一个比对方案就是一条从左上角格子移动到右下角格子的路径,一个矩阵的路径包含了两条序列间所有可能的比对情况,我们要做的就是找出这其中的最优的路径,这其中的方法就是根据分值矩阵来搜索。这里我们假设想要比对的序列分别为seq1和seq2:
>seq1
TCATC
>seq2
TCATGGC在计算矩阵的时候,需要在两条序列之前加上一个 “-”,表示gap。左上角的格子为起点,记为0。接下来看矩阵里剩下的格子,每个格子都由别的格子推算而来,用 C(i, j) 表示第 i 行,第 j 列的格子的得分,计算方法:

其实对应的就是从“上”,”对角线“,”左“ 三个方向分别计算分值,取最大值。第一行和第一列的意义有点特殊,表示序列的起始阶段比对到gap上,因此这里的打分就是开 1 个gap,扣1 分,gap越多,扣的越多。

接下来就是剩下的格子,这就需要比较三个方向计算出的最大值了。以下图为例,要计算T对T这个格子的分值,首先T对T自身是一个完美匹配,+1,从对角线而来就是0+1,得到1;从上面来就是-1-1,得-2;从左边来就是-1-1,得-2。显然取最大值1,同时我们知道了这个T-T位置的1是来自对角线的。
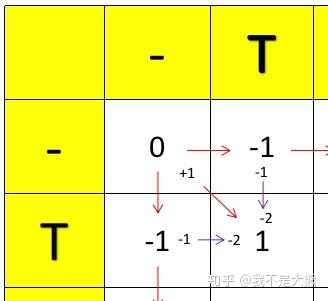
剩下的所有格子都可以此类推,每到一个格子干两件事:计算分值,记录路径:

最终计算到了右下角,这里值得注意的是从最左上角的根出发,整个路径其实是一颗树,只有一条能够到达最右下角(严格来说并不是唯一的,这里先不考虑),因此引出接下来的第三步:
第三步,矩阵生成完之后,开始回溯(traceback)。我们需要知道到底最右下角的值是经历怎样的路径计算而来,这路径就是我们需要的比对结果。
——————————————————— 下篇 ————————————————————
关于Python代码部分,我先做几点说明,考虑到本文定位比较基础,所以:1,不尝试优化代码执行效率,只展示算法逻辑;2,仅使用Python内置的数据类型;3,采用简单的函数式编程,不采用OOP。下面就开始撸代码:
首先,搭个架子:
def main():
pass
return
if _name == '__main__':
main()我们定义一个 theta() 函数来实现任意两个碱基之间的打分:
def theta(a, b):
if a == '-' or b == '-' or a != b: # gap or mismatch
return -1
elif a == b: # match
return 1接下来,算打分矩阵。为了方便,这里使用双层字典来构造矩阵,并且同时构造两个矩阵,一个是score_mat,用来记录每个格子的分值;一个是trace_mat,用来记录相应格子里的值是由那个方向计算而来(以后回溯会用到),我们约定:0表示从对角线来,也就是左上角,1表示从左边来,2表示从上面来:
def make_score_matrix(seq1, seq2):
"""
return score matrix and map(each score from which direction)
0: diagnosis
1: up
2: left
"""
seq1 = '-' + seq1
seq2 = '-' + seq2
score_mat = {}
trace_mat = {}
for i,p in enumerate(seq1):
score_mat[i] = {}
trace_mat[i] = {}
for j,q in enumerate(seq2):
if i == 0: # first row, gap in seq1
score_mat[i][j] = -j
trace_mat[i][j] = 1
continue
if j == 0: # first column, gap in seq2
score_mat[i][j] = -i
trace_mat[i][j] = 2
continue
ul = score_mat[i-1][j-1] + theta(p, q) # from up-left, mark 0
l = score_mat[i][j-1] + theta('-', q) # from left, mark 1, gap in seq1
u = score_mat[i-1][j] + theta(p, '-') # from up, mark 2, gap in seq2
picked = max([ul,l,u])
score_mat[i][j] = picked
trace_mat[i][j] = [ul, l, u].index(picked) # record which direction
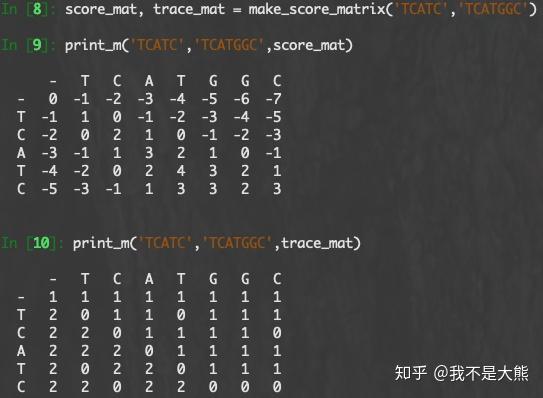
return score_mat, trace_mat写一个辅助函数用来打印矩阵,看看效果(主要为了自己学习和调试用)。这个函数打印 score_mat 和 trace_mat 都可以:
def print_m(seq1, seq2, m):
"""print score matrix or trace matrix"""
seq1 = '-' + seq1; seq2 = '-' + seq2
print()
print(' '.join(['%3s' % i for i in ' '+seq2]))
for i, p in enumerate(seq1):
line = [p] + [m[i][j] for j in range(len(seq2))]
print(' '.join(['%3s' % i for i in line]))
print()
return运行一下试试(截图的显示效果比较理想):
接下来就是准备回溯,寻找”比对解“了。
但立即就遇到一个现实问题:即便知道了如何寻找比对解,知道了路径,以什么形式返回呢,也就是如何记录比对解呢?这里我采用办法是用一段字符串记录比对解,即 path_code。
path_code是一个字符串,从左至右记录了打分矩阵中路径从左上角起点到右下角终点是如何移动的,0表示沿对角线走,1表示往下走(在seq2上开gap),2表示往右走(在seq1上开gap)。
不忙着寻找path_code,我们先来看看如何根据 path_code 打印比对结果:
def pretty_print_align(seq1, seq2, path_code):
'''
return pair alignment result string from
path code: 0 for match, 1 for gap in seq1, 2 for gap in seq2
'''
align1 = ''
middle = ''
align2 = ''
for p in path_code:
if p == '0':
align1 = align1 + seq1[0]
align2 = align2 + seq2[0]
if seq1[0] == seq2[0]:
middle = middle + '|'
else:
middle = middle + ' '
seq1 = seq1[1:]
seq2 = seq2[1:]
elif p == '1':
align1 = align1 + '-'
align2 = align2 + seq2[0]
middle = middle + ' '
seq2 = seq2[1:]
elif p == '2':
align1 = align1 + seq1[0]
align2 = align2 + '-'
middle = middle + ' '
seq1 = seq1[1:]
print('Alignment:\n\n ' + align1 + '\n ' + middle + '\n ' + align2 + '\n')
return运行效果:

那么接下来就只剩最后一个最难,也是最核心的问题了,寻找“比对解”,也就是回溯(trace back)。
这里必须要介绍动态规划算法了。总体上来说,动态规划算法就是把一个大问题分解为一个个可重复的小问题,然后一个一个解决小问题,也就解决了整个大问题。
这里有两个关键点:1,整个问题可以拆分为若干个相似的小问题,能够循环;2, 循环有终点。我们的回溯问题就是一个动态规划问题:从最右下角出发,每到一个格子,查找他的源格子,移动到源格子,再查找源格子的源格子,移动到源格子的源格子,以此类推,直到回到矩阵最左上角的根。记录这一路走来的路径就是答案了,也就是path_code。
def traceback(seq1, seq2, trace_mat):
'''
find one optimal traceback path from trace matrix, return path code
-!- CAUTIOUS: if multiple equally possible path exits, only return one of them -!-
'''
seq1, seq2 = '-' + seq1, '-' + seq2
i, j = len(seq1) - 1, len(seq2) - 1
path_code = ''
while i > 0 or j > 0:
direction = trace_mat[i][j]
if direction == 0: # from up-left direction
i = i-1
j = j-1
path_code = '0' + path_code
elif direction == 1: # from left
j = j-1
path_code = '1' + path_code
elif direction == 2: # from up
i = i-1
path_code = '2' + path_code
return path_code至此整个问题就都解决了,完整可运行的脚本:
import sys
def theta(a, b):
if a == '-' or b == '-' or a != b: # gap or mismatch
return -1
elif a == b: # match
return 1
def make_score_matrix(seq1, seq2):
"""
return score matrix and map(each score from which direction)
0: diagnosis
1: up
2: left
"""
seq1 = '-' + seq1
seq2 = '-' + seq2
score_mat = {}
trace_mat = {}
for i,p in enumerate(seq1):
score_mat[i] = {}
trace_mat[i] = {}
for j,q in enumerate(seq2):
if i == 0: # first row, gap in seq1
score_mat[i][j] = -j
trace_mat[i][j] = 1
continue
if j == 0: # first column, gap in seq2
score_mat[i][j] = -i
trace_mat[i][j] = 2
continue
ul = score_mat[i-1][j-1] + theta(p, q) # from up-left, mark 0
l = score_mat[i][j-1] + theta('-', q) # from left, mark 1, gap in seq1
u = score_mat[i-1][j] + theta(p, '-') # from up, mark 2, gap in seq2
picked = max([ul,l,u])
score_mat[i][j] = picked
trace_mat[i][j] = [ul, l, u].index(picked) # record which direction
return score_mat, trace_mat
def traceback(seq1, seq2, trace_mat):
'''
find one optimal traceback path from trace matrix, return path code
-!- CAUTIOUS: if multiple equally possible path exits, only return one of them -!-
'''
seq1, seq2 = '-' + seq1, '-' + seq2
i, j = len(seq1) - 1, len(seq2) - 1
path_code = ''
while i > 0 or j > 0:
direction = trace_mat[i][j]
if direction == 0: # from up-left direction
i = i-1
j = j-1
path_code = '0' + path_code
elif direction == 1: # from left
j = j-1
path_code = '1' + path_code
elif direction == 2: # from up
i = i-1
path_code = '2' + path_code
return path_code
def print_m(seq1, seq2, m):
"""print score matrix or trace matrix"""
seq1 = '-' + seq1; seq2 = '-' + seq2
print()
print(' '.join(['%3s' % i for i in ' '+seq2]))
for i, p in enumerate(seq1):
line = [p] + [m[i][j] for j in range(len(seq2))]
print(' '.join(['%3s' % i for i in line]))
print()
return
def pretty_print_align(seq1, seq2, path_code):
'''
return pair alignment result string from
path code: 0 for match, 1 for gap in seq1, 2 for gap in seq2
'''
align1 = ''
middle = ''
align2 = ''
for p in path_code:
if p == '0':
align1 = align1 + seq1[0]
align2 = align2 + seq2[0]
if seq1[0] == seq2[0]:
middle = middle + '|'
else:
middle = middle + ' '
seq1 = seq1[1:]
seq2 = seq2[1:]
elif p == '1':
align1 = align1 + '-'
align2 = align2 + seq2[0]
middle = middle + ' '
seq2 = seq2[1:]
elif p == '2':
align1 = align1 + seq1[0]
align2 = align2 + '-'
middle = middle + ' '
seq1 = seq1[1:]
print('Alignment:\n\n ' + align1 + '\n ' + middle + '\n ' + align2 + '\n')
return
def usage():
print('Usage:\n\tpython nwAligner.py seq1 seq2\n')
return
def main():
try:
seq1, seq2 = map(str.upper, sys.argv[1:3])
except:
seq1, seq2 = 'TCATC','TCATGGC'
usage()
print('--------Demo:-------\n')
print('1: %s' % seq1)
print('2: %s' % seq2)
score_mat, trace_mat = make_score_matrix(seq1, seq2)
#print_m(seq1, seq2, score_mat)
#print_m(seq1, seq2, trace_mat)
path_code = traceback(seq1, seq2, trace_mat)
pretty_print_align(seq1, seq2, path_code)
#print(' '+path_code)
if __name__ == '__main__':
main()运行效果:
如果不加任何参数,运行帮助信息:

常规使用,直接接上两条序列:

代码开源在:
希望能对大家有所帮助,如有不当之处,还望大家留言评论或私信我。
———————————————— 20191120 终 ——————————————————


