蛋白质互作网络分析总结
蛋白质互作网络是由蛋白通过彼此之间的相互作用构成,来参与生物信号传递、基因表达调节、能量和物质代谢及细胞周期调控等生命过程的各个环节。
系统分析大量蛋白在生物系统中的相互作用关系,对了解生物系统中蛋白质的工作原理,了解疾病等特殊生理状态下生物信号和能量物质代谢的反应机制,以及了解蛋白之间的功能联系都有重要意义。

那么如何分析蛋白质互作网络?
如何发现复杂网络的关键蛋白和子网络?
如何使用马尔科夫(MCL)聚类算法对功能类似的蛋白进行聚类?
Cytoscape绘制蛋白质互作网络
String (https://string-db.org/)是在线分析蛋白相互作用的数据库,包含5090个物种、24584628个蛋白质与3123056667种相互作用,目前最新版本为2019年1月19日发布的String 11.0。

String 11.0搜索界面简洁明了,可按多种方式进行检索:

例如按名称搜索(Protein by name),输入蛋白名c1orf95,选择物种为Homo sapiens:

点击Search即可得到c1orf95的蛋白互作网络,此时c1orf95处于核心位置(红色节点):
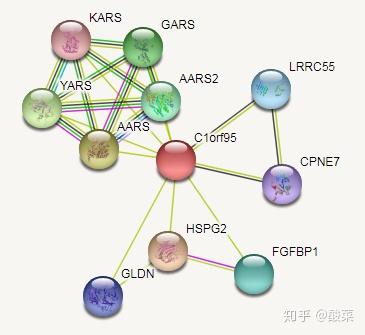
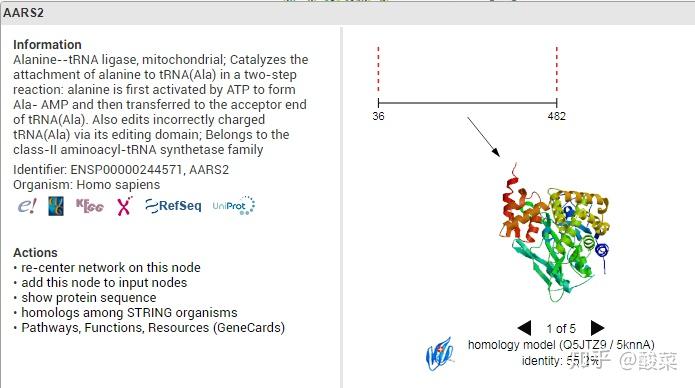
点击圆圈节点可显示该蛋白的具体信息,例如点击AARS2,其信息如下:
Legend(图例)处有对互作网络图中标识的详细解释:

如想查看更多与c1orf95形成互作网络的蛋白可以点击上图More即可得到更复杂的网络,同理点击Less会使网络节点减少。下图是点击More后的互作网络:

在Analysis可对网络进行功能富集,包括Biological Process (GO)、Molecular Function (GO)、Cellular Component (GO)、KEGG Pathways与Reactome Pathways等:


通常情况下我们需要对差异蛋白进行互作网络分析以了解研究背后的蛋白组基础。此时我们需要选择查询Multiple proteins,输入差异表达蛋白,选择物种点击Search:

可以得到输入差异蛋白的蛋白质网络图,可以看到并不是所有差异蛋白都可以形成互作网络:

如果以上图作为差异蛋白互作网络不够美观,也并不是所有差异蛋白均可形成互作网络,因此需将分析结果导出使用Cytoscape绘图。
点击Exports,选择…as simple tabular text output,点击download:

Excel表格打开保存好的结果,其中包括节点信息、node1_string_internal_id与combined_score等诸多信息:

是时候该下一个主角Cytoscape上场了!
Cytoscape是一款免费图形化显示网络并进行分析和编辑的软件,下载地址为:https://cytoscape.org/,其最新版本为Cytoscape3.7.2:

步骤:
1.导入互作网络
打开安装好的Cytoscape软件:

点击下图Import Networks from File System,导入Exports处下载好的数据:

导入完成后得到可形成互作关系的差异蛋白网络,不能形成互作关系的单个差异蛋白不再出现:

2.设置节点间连接线的粗细
combined_score表示数据的支持度,combined_score越粗两种蛋白间互作关系越强,默认筛选阈值为0.4。
节点Edges的粗细由combined_score渐变调整,选择Tool里的Network Analysis工具中的Generate Style from Statistics:

不同节点之间连接线的粗细发生了改变:

上图背景为灰色,点击Network -> Background Paint,设置背景颜色为纯白(R,G,B,255,255,255):

3.设置Nodes的大小与颜色
与之形成相互作用的蛋白节点数量越多,蛋白Nodes越大。此时需要使用Excel中对指定区域中符合指定条件的单元格计数的一个函数Countif。
将Nodes1和Nodes2复制到一列中,然后利用=COUNTIF(A:A,A2)的公式进行计算其出现频数:

删除第一列中的重复值,方法为Excel表格中数据下的重复值删除:

添加一列与Nodes1相同的加一列Nodes2:
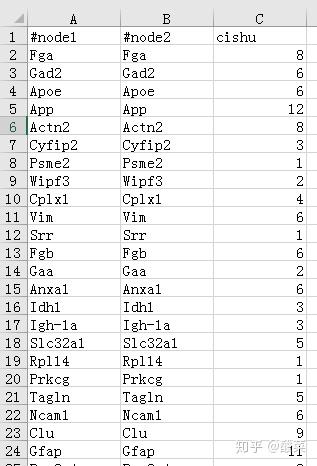
根据蛋白质组学添加上下调关系(1代表上调,2代表下调):
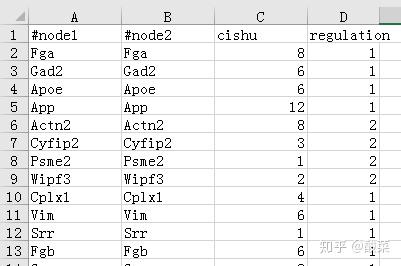
将上述表格保存为txt文件:
File -> import -> Table from file导入Cytoscape软件:

由cishu决定Nodes大小,Mapping Type选择Continuous Mapping:
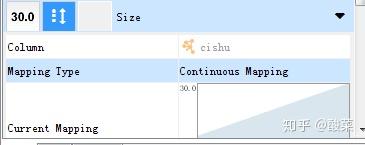
此时节点显示为不同的大小:

Fill Color,颜色由Column,Regulation决定,上调设置为红色,下调设置为绿色:
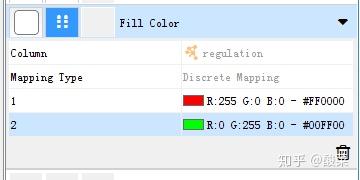
此时节点显示为不同的颜色,红色代表上调、绿色代表下调:

鼠标拖动节点使蛋白互作网络紧凑、美观:
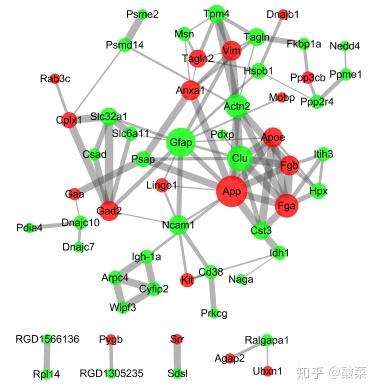
4.导出互作网络图
File -> Export -> Network to Image导出图片即可:

筛选互作网络的关键蛋白和子网络
在复杂的差异蛋白互作网络中获取关键蛋白和子网络对于寻找生命活动的机制显得尤为重要。

CytoHubba插件介绍
Cytoscape有诸多使用插件,其中cytoHubba插件可以通过几种拓扑算法预测和探索给定网络中的关键节点和子网络。
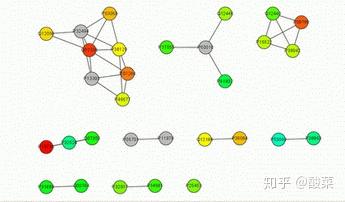
cytoHubba提供了11种拓扑分析方法,包括Degree, Edge Percolated Component (EPC), Maximum Neighborhood Component (MNC), Density of Maximum Neighborhood Component (DMNC), Maximal Clique Centrality (MCC) 等。
一般而言具有高degree的蛋白更倾向于是关键蛋白,MCC在酵母互作网络中对关键蛋白的预测有更好的表现。
CytoHubba插件安装
打开Cytoscape软件,点击Apps,进入App manager:
在App manager中搜索cytoHubba,点击Install进行安装,安装好的插件可在Apps处显示:

CytoHubba插件的使用
1.打开绘制好的差异蛋白互作网络图

2.点击cytoHubba,Target Network选择打开的网络,点击Calculate:

默认设置显示Top10的蛋白节点,如需显示所有形成网络的节点则填入58。排序的拓扑分析方法有多种,选择degree:

得到Top 10蛋白节点,右侧是蛋白节点排序,颜色越深蛋白在互作网络中越重要:

点击Save Current Rank保存当前排序:
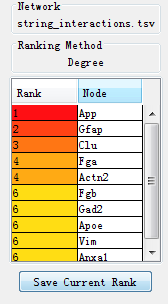

Top 58显示所有蛋白节点排序:

美化网络后导出图片即可:

对功能类似的蛋白进行MCL聚类
下表是文献中对上调蛋白进行MCL聚类后进行的GO分析:

其MCL聚类互作网络图为:

那么如何对功能类似的蛋白进行MCL聚类呢?
首先,打开String 11.0,输入待查询差异蛋白:

得到互作网络图:

点击Clusters,选择MCL Clustering -> APPLY:

在Save / Export下可以下载MCL聚类结果:

结果显示MCL可以聚类为几类,每类有几个蛋白,以及参与聚类的具体蛋白。
例如聚类1有8个蛋白组成,标记颜色为红色:

后续可以利用Cytoscape可以进行每个具体的聚类的互作网络图:

转载请注明:解螺旋·临床医生科研成长平台。
点击下面的链接可扫码添加我的助理微信,私信不回问题:
传播科研最前沿知识;
中国临床医生科研成长平台;
现在添加还可领取10G科研资源大礼包!(内含:SCI写作投稿技巧、数据库使用教程、国自然工具包……)

