R_语言 ENSG-GeneSymbol-GeneID转换
在TCGA数据下载后,得到的常常是ENSG号,而我们在文章中常用到的是genesymbol和/或geneid,如何转换呢?代码如下
首先需要普及的一个问题,我们下载的ENSG数据中常常有小数点,小数点后面的表示的版本类型,所有,在进行匹配时需要删除小数点后面的,一般情况下ENSG一共是15位,所有保留前15位字符就可以了。
##read.delim()函数读取tsv数据
exp <- read.delim("TCGA-BRCA.htseq_counts.tsv",stringsAsFactors=FALSE)
data=data.frame(exp)library(stringi)##加载包
data$Ensembl_ID=stri_sub(data$Ensembl_ID,1,15)##保留前15位
# 加载相关包
library(clusterProfiler)
library(org.Hs.eg.db)
# 查看org.Hs.eg.db 包提供的转换类型
keytypes(org.Hs.eg.db)
# 需要转换的Ensembl_ID
Ensembl_ID <- data$Ensembl_ID
# 采用bitr()函数进行转换
gene_symbol <- bitr(Ensembl_ID, fromType="ENSEMBL", toType=c("SYMBOL", "ENTREZID"), OrgDb="org.Hs.eg.db")
# 查看转换的结果
head(gene_symbol)data=data.frame(gene_id,data[match(gene_id$ENSEMBL,data$Ensembl_ID),])#匹配到表达矩阵中
data=data[,-4]#去除重复的Ensembl_ID列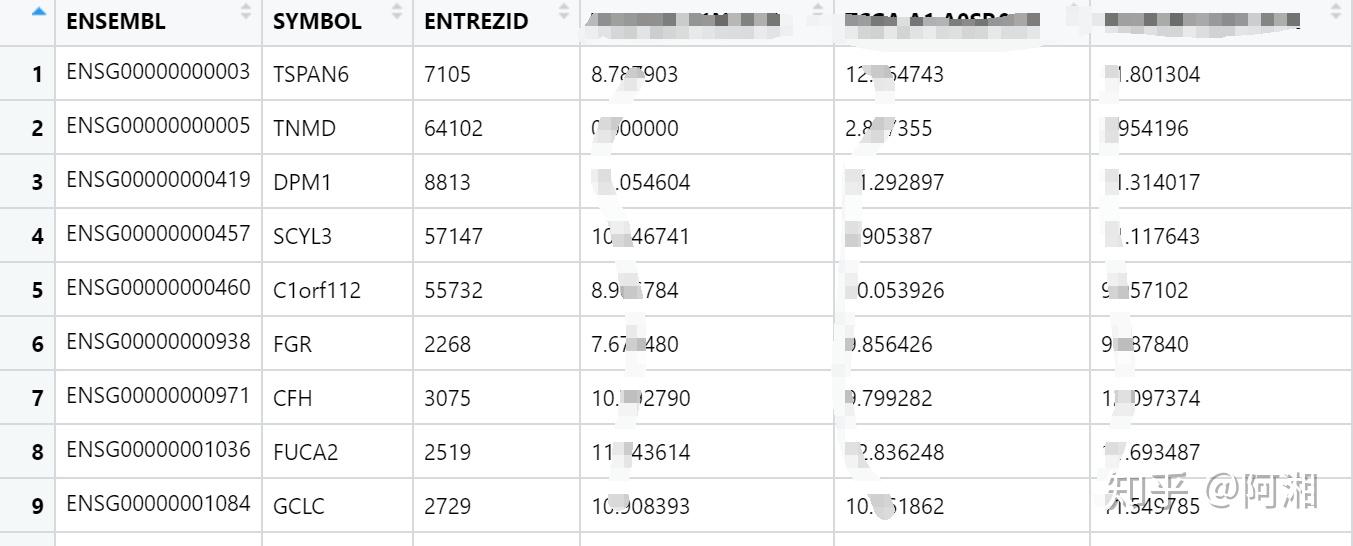
发布于 2020-05-05 00:03
